來源:藥明康德
就在人工智能學者出人意料地摘得2024年諾貝爾物理學獎之后,北京時間10月9日下午,諾貝爾基金會宣布將今年的諾貝爾化學獎頒發給了三位研究領域與人工智能相關的科學家。
其中,被譽為“AlphaFold之父”的谷歌DeepMind公司Demis Hassabis博士和John Jumper博士因為蛋白質結構預測獲獎,而華盛頓大學的David Baker教授因計算蛋白設計榮獲殊榮。他們開發的AI解決方案成功解決了50年來蛋白質結構預測領域的重大挑戰,加速了生物醫藥領域的科學發現。

下面,藥明康德內容團隊將與大家一道回顧這個革命性的AI解決方案的誕生和成長史。
首先,讓我們來看兩位共同主導了AlphaFold開發的獲獎者的故事。其中,Demis Hassabis博士是DeepMind的創始人兼首席執行官,同時也是AlphaFold項目的負責人。John Jumper博士則是AlphaFold項目的首席高級研究員。我們都知道,蛋白質是維持我們生命所必需的龐大而復雜的物質。我們身體的幾乎所有功能,例如收縮肌肉、感知光線或將食物轉化成能量等,都需要一種或多種蛋白質來完成。而蛋白質具體能做什么就要取決于它獨特的3D結構了。然而,純粹基于其基因序列推測蛋白質的3D結構是一項非常具有挑戰性的復雜任務。這是因為我們的DNA通常只包含蛋白質中氨基酸殘基的序列信息,而這些氨基酸殘基形成的長鏈將會折疊成錯綜復雜的3D結構,加上蛋白質越大,需要考慮的氨基酸之間的相互作用就越多,對其結構的建模過程就會更加復雜和困難。在過去的數十年中,科學家們已經能夠利用冷凍電子顯微鏡、核磁共振或X射線晶體學等技術在實驗室中確定蛋白質的形狀,但這些方法都需要通過大量的試錯才能獲得最終的結果,這可能需要花上好幾年時間以及大量的資金。幸運的是,得益于基因測序成本的快速降低,基因組學領域的數據變得豐富了起來。一些科學家們開始利用AI技術開發深度學習算法,在基因組學數據的基礎上對蛋白質結構進行預測。在此基礎上,AlphaFold誕生了。
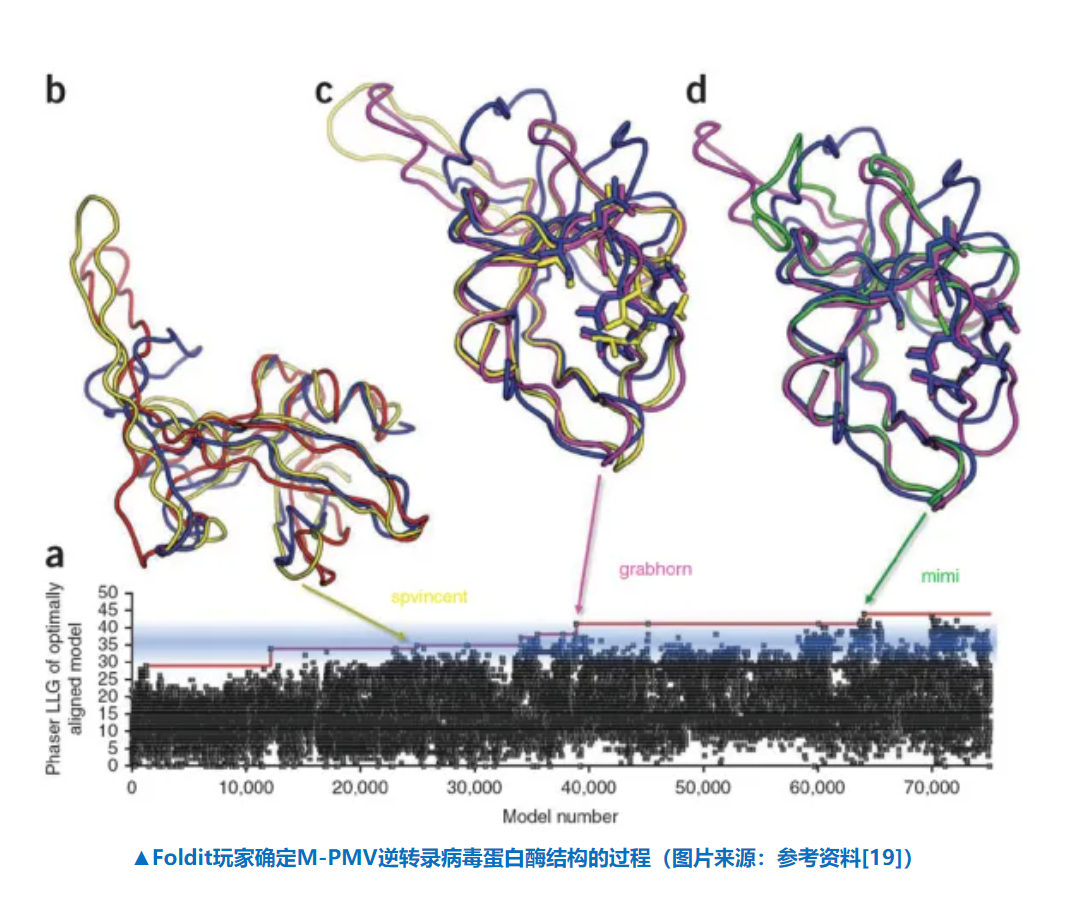
2018年12月,DeepMind宣布推出全新的AlphaFold系統,能夠預測并生成蛋白質的3D結構。在當年的國際蛋白質結構預測競賽(CASP)上,初次登場的AlphaFold就成為了最大的黑馬,以絕對的優勢擊敗了上百位的參會選手,拔得頭籌。在比賽中,AlphaFold成功預測了給定的43種蛋白質中的25種的最準確結構,而同一類別的第二名參賽隊伍只預測出了43種中的3種。和以往依賴預先構想邏輯的傳統人工智能方法不同的是,AlphaFold并未使用已經明確結構的蛋白質3D模型作為模板,而是通過將機器學習作為蛋白質結構預測網絡的核心組成部分,讓AlphaFold從數據中自行發現模式規律。DeepMind團隊使用的方法都以深度神經網絡為基礎,從基因序列中預測蛋白質的兩種物理性質:氨基酸對之間的距離及連接這些氨基酸的化學鍵之間的角度。首先,研究小組訓練了一個深度神經網絡,來預測蛋白質中每對氨基酸殘基之間距離的分布情況。然后,研究人員將這些數值轉化為評分,來對蛋白質結構的精確程度進行評估。同時,研究人員還另外訓練了一個神經網絡,利用這些距離數值來評估預測結構與真實結構的接近程度。不僅如此,DeepMind的研究人員還在這些評分函數的基礎上,使用了兩種全新的方式來優化蛋白質結構評分:他們使用了一個生成神經網絡,不斷生成新的蛋白質片段來反復替換一段舊的蛋白質結構,這樣一來,蛋白質結構的評分就被不斷提高了。另外,研究人員還使用了一種名為梯度下降的方式來讓AlphaFold預測的結構變得高度精確。梯度下降是一種機器學習中常用的數學技術,用來實現漸進式的細微改進。研究人員將這項技術用于整個蛋白質鏈,而不是結構中組裝前必須分開折疊的片段,降低了預測過程的復雜性。盡管AlphaFold的首戰告捷,但DeepMind的研究人員并不滿意:他們希望得到一種對于實驗人員更加有用的工具,誤差小于1埃米(原子的大小)。經過多輪的調試和集思廣益,DeepMind的研究團隊在原來的算法基礎上成功構建出了AlphaFold2。在2020年的CASP上,DeepMind的AlphaFold2系統表現驚艷,在接受檢驗的近100個蛋白靶點中,AlphaFold2對三分之二的蛋白靶點給出的預測結構與實驗手段獲得的結構相差無幾。有些情況下,已經無法區分兩者之間的區別是由于AlphaFold2的預測出現錯誤,還是實驗手段產生的假象。2021年,Hassabis博士和Jumper博士與歐洲分子生物學實驗室的歐洲生物信息學研究所(EMBL-EBI)合作,發布了AlphaFold預測的蛋白結構數據庫(AlphaFold Protein Structure Database)。這個數據庫涵蓋了人類和20種常用模式生物的35萬個蛋白質結構,并且對98.5%的人類蛋白質結構進行了準確預測——要知道在此之前,科學界解析的蛋白質結構只覆蓋了人類蛋白序列17%的氨基酸。歐洲生物信息研究所主任Ewan Birney博士稱該數據庫為人類基因組圖譜發布以來最重要的數據庫之一。人工智能預測蛋白質結構領域的一系列突破,也被《科學》評選為2021年的年度科學突破。而更令人激動的是,他們開發的這一數據庫將免費提供給全球的科研人員開放使用!許多科學家和生物醫藥公司的研究員興奮地表示,這一系列突破將加速新藥開發,并為基礎科學帶來全新革命。2022年,DeepMind與EMBL-EBI團隊的合作又迎來了一項巨大的飛躍。AlphaFold對蛋白質結構的預測不再局限于人類與模式生物,而是拓展至涵蓋了動植物、細菌等的100萬個物種。不僅如此,其預測的蛋白質結構數量也提升了數百倍。AlphaFold2已對超過2億種蛋白質進行了結構預測——幾乎是科學界已知的所有蛋白質。同樣的,這2億種蛋白質的結構預測數據依然向公眾免費開放,使研究人員能夠像使用谷歌搜索信息一樣搜索蛋白質的結構,為研究人員即時提供他們正在研究的任何蛋白質的預測模型,大大減少了他們曾經需要花在確定蛋白質結構上的時間。目前,這些數據已經在瘧疾疫苗開發、解決抗生素耐藥性問題與塑料污染等場景中得到應用,并能夠幫助研發人員加速新藥研發。除此以外,該模型還具有加速生物學各個研究領域的潛力,其應用前景正等著更多才華橫溢的科學家們來盡情開發。提到蛋白質從頭設計,華盛頓大學蛋白質設計研究所所長David Baker博士的大名可謂是無人不知。不過,可能很少有人知道,在走上生物化學研究的道路前,Baker的專業是研究哲學。1983年,一堂關于蛋白質折疊問題的生物學課程徹底改變了他的人生軌跡。自那以后,這個眾多科學家前赴后繼嘗試破解的生物學難題便成了他畢生的研究課題。在生物體內,蛋白質讓很多科學家們著迷。這種分子的尺寸只有納米大小,復雜程度卻可以超過任何一臺人造的機器,大自然的精妙由此也可見一斑。1983年,在哈佛大學學習哲學的Baker在一堂生物學課程上了解到蛋白質折疊問題。此前的科學家們通過試驗發現,這些復雜的蛋白質只由20種簡單的氨基酸經過排列組合拼接而成,而一條氨基酸序列就已包含了它能形成蛋白質的所有結構和活性信息。就像有設計圖紙一樣,一條氨基酸序列可以自發折疊成唯一的三維結構,然后在細胞內發揮特定的功能——有的可以結合DNA,控制基因的開關;有的可以識別病原體,啟動免疫反應。在這些現象背后,一個巨大的問題隨之浮現:一條氨基酸序列從理論上來說可以有無數種折疊方式,那為什么它能夠自發折疊成唯一的三維結構呢?自那堂課后,Baker對這個數十年來困擾了無數科學家的難題產生了極大的興趣,甚至不惜轉換專業在生物學領域從頭開始學習。而當他和導師提起他想要對這個難題發起挑戰時,他的導師勸他不要頭腦發熱,因為“沒人知道這是怎么回事”。聽從了導師的建議,Baker將這一念頭短暫封存,并在未來的諾獎得主Randy Schekman教授課題組獲得了博士學位,主攻細胞生物學。博士后期間,Baker接觸到使用計算機科學來進行結構生物學研究的方法。在這個過程中,他發現使用計算機解析晶體結構并不是他擅長的,但他卻萌生出了另一個想法,或許計算機可以幫他實現那個他始終放不下的夢想——解開蛋白質折疊之謎。1993年,Baker成功獲得了華盛頓大學生物化學系助理教授的職位,開始獨立工作。在他招收第二個學生后,他建議學生借助計算機的力量做蛋白質結構預測相關的課題。1996年,他與研究生們開始編寫一個叫做Rosetta的程序,這個程序有潛力根據一段氨基酸序列解出蛋白質的結構。在自然界中,為了保持穩定,蛋白質總是折疊成具有“最低自由能”的形狀。這就好像水會從高處往低處流,然后停留在那里一樣。不過利用計算機預測蛋白質結構也并沒有想象中那么簡單。由于每個氨基酸至少有三種不同的構象,那么一個僅含有100個氨基酸的蛋白質,其可能的結構就高達3的100次方種,這對計算機來說都是個難以處理的運算量。不過,Rosetta的程序設計用了一種十分巧妙的方法,它不是通過窮舉法從這些天文數字般的可能結構中挨個尋找自由能最低的形狀,而是先分析蛋白質的生物物理特性,模擬出一個大致的形狀,然后進行微調,只留下自由能更低的結果。這樣一來,研究人員們可以更快預測出蛋白質的結構。好消息是,Rosetta的表現十分驚艷。自1994年起,和Baker一樣想要解開蛋白折疊之謎的生物學家們會定期聚在一起,檢驗各自的成果:就像考試一般,他們會拿到一個蛋白質的序列,然后預測出它的結構。隨后,這些預測結構會和已通過實驗方法得到解析但尚未公開的真實結構進行比對,看哪一個結構更為接近。在這個被譽為蛋白質結構領域“奧林匹克”的活動中,Rosetta程序總是最有力的競爭者,并且具有統治性的優勢。在Rosetta誕生的過程中,Baker還有許多意料之外的收獲。盡管Rosetta的設計經過優化,但預測蛋白質折疊所需要的運算量依然巨大。最開始,Baker只能通過不停購置新的電腦設備來擴大計算力,后來,新買的電腦把實驗室的空間占滿了卻依然無法滿足他們的需求。迫于這樣的壓力,Baker和他的學生們想出了一個絕妙的解決方案——借助互聯網,邀請世界各地的人們用他們計算機的閑置算力來幫助進行計算。2005年,Baker團隊啟動了一個名為Rosetta@home的項目,基于他們開發的Rosetta軟件包,利用分布式計算的力量來解析蛋白質結構。令人感到意外的是,這些“網友”們還給Baker發去了反饋意見,表示計算機折疊沒有他們手動折疊來得更好。更巧的是,當他與一名計算機科學家聊起這些話題時,倆人靈感迸發,決定從Rosetta@home出發開發一款游戲,讓全世界對蛋白折疊感興趣的人能夠發揮他們的才華,參與到蛋白質折疊的解謎游戲中。這款名為Foldit的游戲由于能幫助學生更好地了解蛋白質的三維性質以及蛋白質結構和功能間的關系,已被一些大學引入課堂。更令人吃驚的是,一些該游戲的高級玩家還曾通過這款游戲破解了一種逆轉錄病毒的蛋白結構,并將成果發表在了《自然》雜志子刊上。
除此以外,與Foldit同時期誕生的還有一個名為Rosetta Commons的學術團體。這個團體的成員包括許多高校和研究機構的人員,其中很多都在Baker的實驗室工作過。除了日常的交流合作,他們會定期舉辦會議分享最新成果、討論如何進一步優化Rosetta,并開設訓練營培訓那些對Rosetta感興趣但不知道如何使用的人。雖然Baker最初的研究方向是預測蛋白質的結構,但在這個方向上取得突破之前,他已著手向另一個截然相反且更具挑戰性的領域——“蛋白質的從頭設計”發起了沖擊。相比于預測蛋白質的結構,從頭設計出一個蛋白質需要向弄清蛋白質折疊的原理再邁進一步。這要求科學家們能根據一個具有特定形狀的蛋白,倒推出其DNA序列。從某種意義上講,從頭設計蛋白,要比預測蛋白結構難上幾個數量級。假設要設計一個由100個氨基酸組成的蛋白質,每一種氨基酸又有20種截然不同的可能,使將得可能的氨基酸序列總數高達20的100次方。這個數字究竟有多大?它比整個宇宙中原子的總數還要多!由于Baker在Rosetta的開發中已經取得過一定的經驗,這次再開發從頭設計蛋白質的方法就有了良好的基礎。從DNA序列到蛋白質結構,Rosetta能找到能量最低的形狀。那么反過來,Rosetta也能用來推導為了構成這一形狀所需的蛋白組件。在此基礎上,研究人員們還學會了如何像拆解樂高玩具一樣,將一個蛋白質拆成螺旋或者桶裝的小塊,分塊擊破。
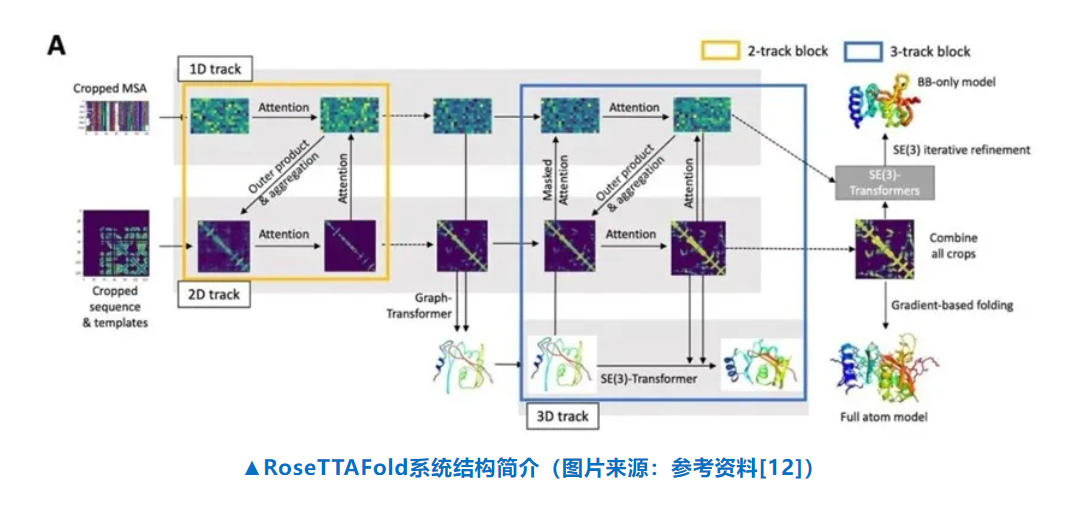
2003年,Baker的團隊設計出了第一個原本并不存在于自然界中的蛋白質,它被命名為Top7。這當然是一個重要突破,但卻沒有開辟一個嶄新的時代。Baker實驗室的成員開玩笑說Top7只是一塊從熱力學角度上看很穩定的“石頭”。因為他們從頭設計出的這個蛋白質雖然折疊成了研究人員們想要它折疊的模樣,但不具有任何功能。7年后,Baker的一名博士后研究員做出了改進。他將抗體的一部分連接到了人造蛋白上,使人造蛋白首度具有了功能:新合成的蛋白能識別流感病毒,有望成為一種新的藥物,但這多少有些“作弊”的意思,畢竟最重要的那部分來自天然的抗體。接下來的幾年時間,Baker的團隊對Rosetta進行了更多的優化。如今,Baker的實驗室,以及他的合作伙伴們已能設計出多種不同的蛋白,有朝一日,人類完全獲得“上帝之手”的能力將不再是夢想。不過到目前,從頭設計蛋白依然是一個不斷試錯的工作,需要大量的資源投入。以設計結合蛋白為例,從流程上看,科學家會首先用Rosetta模擬出所感興趣蛋白表面上的一個“口袋”,然后再設計出大量不同的螺旋結構,形成穩定骨架。這些骨架上含有一些特定的氨基酸,有可能會與“口袋”進行完美的契合。這個工作就像是在一把鑰匙上不斷打磨,最終使其完美地對應一把鎖。隨后,研究人員們會根據設計合成所要的DNA序列,將其引入細菌細胞,期望它們能夠產生所需要的蛋白。獲取這些蛋白后,他們還會做兩個測試:評估這些蛋白是否能如預期般折疊,以及折疊后的蛋白是否能如預期般結合特定蛋白。通常來講,人工設計的蛋白極少能同時滿足這兩個條件。而那些脫穎而出的蛋白,則會成為新一輪設計與篩選的起點,直至獲得最佳的構象。在2018年以前,Baker及其團隊開發的Rosetta在蛋白質結構預測領域完全沒有對手。而那一年,AlphaFold的出現令Baker嗅到了危機。盡管18年的蛋白質結構預測競賽依然是Rosetta拔得頭籌,但首次亮相就獲得了第二名的AlphaFold令Baker見識到了機器學習的過人之處。于是,他要求團隊緊跟時代的風向,加緊研究機器學習。Baker的預感沒有錯,在2020年的競賽中,第二代AlphaFold擊敗了Rosetta,一舉成名。不過,Baker率領著團隊很快就追趕了上來。2021年7月15日,當DeepMind公司在《自然》雜志上發表論文,公開了“AlphaFold2”的源代碼,并且詳細描述了它的設計框架和訓練方法時,Baker的團隊也于《科學》雜志上介紹了其開發的RoseTTAFold算法。RoseTTAFold的神經網絡能夠同時考慮蛋白序列的模式、蛋白中不同氨基酸之間的相互作用,以及蛋白質可能出現的3D結構。在這個系統中,一維、二維和三維的信息能夠相互交流,讓神經網絡綜合所有信息,決定蛋白質的化學組成部分和它折疊產生的結構之間的關系。
研究人員表示,RoseTTAFold系統在解析蛋白質3D結構方面的表現與AlphaFold2的水平幾乎相當,在有些蛋白上甚至優于AlphaFold2。利用來自AlphaFold的公開信息,也得益于多年來對于機器學習的積累,這個算法的開發只用了區區幾個月的時間。作為蛋白質從頭設計的先驅者,Baker希望通過“蛋白質設計革命”開啟一個全新的時代,我們將學會使用一種前所未有的方式來操控生物分子,例如從頭設計出全新的藥物、疫苗、疾病療法等,拓展新藥研發的邊界。2022年8月,Baker及其團隊在《細胞》雜志上發表論文,他們已利用AI技術平臺精準地從頭設計出能夠穿過細胞膜的大環多肽分子,開辟了設計全新口服藥物的新途徑。同時,Baker團隊成員聯合創建的初創公司Vilya也正式亮相,并從著名風投機構ARCH Venture Partners獲得5000萬美元A輪融資。利用這一技術,跳過高通量篩選、直接合成候選藥物的策略不再遙不可及!今年以來,Baker及其團隊已在《自然》和《科學》雜志上發表了數篇重磅論文,其開發的全新的蛋白質從頭設策略法可靶向不可成藥靶點,并能實現按需設計生物分子,為蛋白設計提供了更廣闊的可能性。
華盛頓大學蛋白設計研究所首席戰略及運營官Lance Stewart博士是David Baker教授長期的合伙伙伴。在2023年藥明康德全球論壇上,他指出:“現在的新技術讓我們有能力去挑戰任何類型的靶點,這是當下生物醫藥產業的幸運。”在獲得此次諾貝爾獎之前,Demis Hassabis博士與John Jumper博士還在今年獲得了多個科學大獎,包括蓋爾德納獎(Gairdner)和拉斯克獎。2020年,Baker博士也獲得了素有“科學界的奧斯卡”之稱的科學突破獎——生命科學科學突破獎。此次共同摘得諾貝爾獎的桂冠是對他們通過放飛想象力和才華,解決了讓科學家困惑了半個世紀的蛋白質結構預測難題的再次肯定。讓我們再次祝賀這三位杰出的科學家,并向他們突出的貢獻致以崇高敬意!
免責聲明:本文系轉載內容,版權歸原作者所有,轉載目的在于傳遞更多信息,并不代表我方觀點。如涉及作品內容、版權和其它問題,請與我方留言聯系,我們將在第一時間刪除內容。